Abstract
目的
恶性黑色素瘤是高度恶性和异质性的皮肤肿瘤,尽管免疫治疗的出现提高了患者的生存率,但肿瘤微环境的抑制作用却减弱了免疫治疗的效果。因此需开发特定的免疫相关的预后模型从而提高患者的生存率和治疗策略。本研究结合癌症基因组图谱(The Cancer Genome Atlas,TCGA)、高通量基因表达(Gene Expression Omnibus,GEO)以及序列读取档案(Sequence Read Archive,SRA)数据库中皮肤黑色素瘤(skin cutaneous melanoma,SKCM)的相关数据,旨在建立一个基于差异表达的免疫相关预后预测模型,并通过风险评分评估肿瘤免疫微环境用以指导临床免疫治疗。
方法
从TCGA数据库获取SKCM转录组测序数据和对应的临床信息,分析其差异表达基因,并使用单因素Cox回归、LASSO方法以及逐步回归建立预后模型。采用实时反转录聚合酶链反应(real-time reverse transcription PCR,real-time RT-PCR)和蛋白质印迹法验证预后模型中基因的表达差异。使用Kaplan-Meier方法进行生存分析,通过时间依赖受试者操作特征曲线以及多因素Cox回归评价模型的效果,并使用GEO数据库中2个SKCM数据集对预后模型进行验证。进一步分析风险评分与免疫细胞浸润、基于表达估计恶性肿瘤组织的基质细胞和免疫细胞(Estimation of STromal and Immune cells in MAlignant Tumor tissues using Expression data,ESTIMATE)评分、免疫检查点mRNA表达水平、肿瘤免疫周期及肿瘤免疫微环境通路间的相关性。最后在真实世界的队列中验证风险评分与免疫治疗效果及预后的相关性。
结果
TCGA-SKCM数据集的差异分析发现差异表达基因主要与肿瘤免疫微环境相关。同时得到一个基于4个基因的预后模型,在模型中SKCM组织与正常皮肤组织的人细胞维甲酸结合蛋白2 (cellular retinoic acid binding protein 2,CRABP2)、白血病抑制因子(leukemia inhibitory factor,LIF)及杀伤细胞凝集素样受体D1(killer cell lectin like receptor D1,KLRD1)基因的mRNA和蛋白质水平差异均有统计学意义(均P<0.01)。高风险评分的患者总生存期显著短于低风险评分的患者(P<0.001),在训练队列与多个验证队列取得了一致的结果(P<0.001)。预后模型与肿瘤免疫微环境的相关性分析结果显示高风险评分的SKCM患者处于抑制性的免疫微环境(P<0.01)。
结论
构建的免疫相关SKCM预后模型可以有效预测SKCM患者的预后;结合其评分与肿瘤免疫微环境的密切相关性,该模型对于SKCM临床免疫治疗的效果预测具有一定的参考价值。
Keywords: 皮肤黑色素瘤, 肿瘤免疫微环境, 预后模型, 生物信息学
Abstract
Objective
Malignant melanoma is a highly malignant and heterogeneous skin cancer. Although immunotherapy has improved survival rates, the inhibitory effect of tumor microenvironment has weakened its efficacy. To improve survival and treatment strategies, we need to develop immune-related prognostic models. Based on the analysis of the Cancer Genome Atlas (TCGA), Gene Expression Omnibus (GEO), and Sequence Read Archive (SRA) database, this study aims to establish an immune-related prognosis prediction model, and to evaluate the tumor immune microenvironment by risk score to guide immunotherapy.
Methods
Skin cutaneous melanoma (SKCM) transcriptome sequencing data and corresponding clinical information were obtained from the TCGA database, differentially expressed genes were analyzed, and prognostic models were developed using univariate Cox regression, the LASSO method, and stepwise regression. Differentially expressed genes in prognostic models confirmed by real-time reverse transcription PCR (real-time RT-PCR) and Western blotting. Survival analysis was performed by using the Kaplan-Meier method, and the effect of the model was evaluated by time-dependent receiver operating characteristic curve as well as multivariate Cox regression, and the prognostic model was validated by 2 GEO melanoma datasets. Furthermore, correlations between risk score and immune cell infiltration, Estimation of STromal and Immune cells in MAlignant Tumor tissues using Expression data (ESTIMATE) score, immune checkpoint mRNA expression levels, tumor immune cycle, or tumor immune micro-environmental pathways were analyzed. Finally, we performed association analysis for risk score and the efficacy of immunotherapy.
Results
We identified 4 genes that were differentially expressed in TCGA-SKCM datasets, which were mainly associated with the tumor immune microenvironment. A prognostic model was also established based on 4 genes. Among 4 genes, the mRNA and protein levels of killer cell lectin like receptor D1 (KLRD1), leukemia inhibitory factor (LIF), and cellular retinoic acid binding protein 2 (CRABP2) genes in melanoma tissues differed significantly from those in normal skin (all P<0.01). The prognostic model was a good predictor of prognosis for patients with SKCM. The patients with high-risk scores had significantly shorter overall survival than those with low-risk scores, and consistent results were achieved in the training cohort and multiple validation cohorts (P<0.001). The risk score was strongly associated with immune cell infiltration, ESTIMATE score, immune checkpoint mRNA expression levels, tumor immune cycle, and tumor immune microenvironmental pathways (P<0.001). The correlation analysis showed that patients with the high-risk scores were in an inhibitory immune microenvironment based on the prognostic model (P<0.01).
Conclusion
The immune-related SKCM prognostic model constructed in this study can effectively predict the prognosis of SKCM patients. Considering its close correlation to the tumor immune microenvironment, the model has some reference value for clinical immunotherapy of SKCM.
Keywords: skin cutaneous melanoma, tumor immune microenvironment, prognostic model, bioinformatics
作为一种高度侵袭性和异质性的恶性肿瘤,黑色素瘤是皮肤癌引起死亡的主要瘤种。早期皮肤黑色素瘤(skin cutaneous melanoma,SKCM)患者常可通过手术治愈,并且有超过90%的5年生存率[1]。而晚期或转移性SKCM通常预后较差,可以选择的治疗手段也非常有限[2]。
肿瘤微环境是肿瘤细胞与宿主免疫系统相互作用的主要场所。除肿瘤细胞外,肿瘤微环境还由多种异质成分组成,包括免疫细胞、基质细胞和细胞外成分,如细胞因子、趋化因子和激素。肿瘤微环境的各种成分不仅在肿瘤进展、免疫逃逸和转移中发挥重要作用,而且对患者的治疗效果也有深远的影响[3-4]。例如,肿瘤微环境内的免疫抑制细胞在促进肿瘤免疫逃逸和通过释放免疫抑制细胞因子的方式促进抗肿瘤免疫反应的局部抑制方面起着关键作用[5]。同样,肿瘤浸润淋巴细胞的表达水平通常与SKCM以及其他实体瘤患者的生存率相关[6-7]。免疫检查点抑制剂(immune checkpoint inhibitor,ICI)作为新兴的免疫治疗策略,极大地提高了各类型癌症患者的生存率[8-11]。然而,免疫治疗对多数肿瘤患者难以长期持续起作用,或者发生免疫治疗耐药反应[12]。免疫治疗的有限效果部分归因于肿瘤微环境的免疫抑制作用。
如何识别免疫治疗的潜在获益者是目前临床重点关注的问题。随着高通量基因测序技术的不断发展,各种大型的肿瘤相关公共数据库不断完善,为探索肿瘤的分子机制以及对患者的分类预测提供了便利。目前尽管不少研究者为SKCM治疗开发了大量预后模型,但存在一定的局限性,如现有的部分SKCM预后模型没有多数据集的广泛验证及临床的初步检测。因此,本研究利用生物信息学方法,基于免疫相关基因,建立一个SKCM的预后模型并通过临床初步验证预后标志物的可靠性,随后通过阐述模型潜在的临床意义和分析其与肿瘤免疫微环境的关联性,旨在揭示预后模型在肿瘤治疗中的预测价值并为寻找预后生物标志物提供更多的证据。
1. 资料与方法
1.1. 数据获取
癌症基因组图谱 (The Cancer Genome Atlas,TCGA)数据库是由美国国家癌症研究所(National Cancer Institute,NCI)和美国国家人类基因组研究所(National Human Genome Research Institute,NHGRI)建立的一个大型肿瘤数据库,其中收录了33种肿瘤,超过20 000例肿瘤和配对正常样本,包括基因组、表观基因组、转录组、蛋白质组数据,以及详细的临床信息。我们从TCGA官网(https://portal.gdc.cancer.gov/)下载TCGA-SKCM数据集的基因组、转录组数据和详细临床信息。
高通量基因表达(Gene Expression Omnibus,GEO)数据库是由美国国家生物技术信息中心(the National Center for Biotechnology Information,NCBI) 创建并维护的基因表达数据库。我们从GEO数据库获取GSE54467和GSE65904SKCM数据集作为验证队列。其中,GSM54467包括79例转移性SKCM患者的转录组数据和临床信息,GSE65904包括214例转移性SKCM患者的转录组数据和临床信息。Gide队列(PRJEB23709:抗PD1单药治疗和抗PD1/抗CTLA4联合治疗)从序列读取档案(Sequence Read Archive,SRA)数据库(https://www.ncbi.nlm.nih.gov/bioproject/)下载。
ImmPort是由美国国立卫生研究院(National Institutes of Health,NIH)、美国过敏症和传染病研究所(National Institute of Allergy and Infectious Diseases,NIAID)和过敏、免疫和移植部(Division of Allergy, Immunology, and Transplantation,DAIT)共同开发维护的一个免疫相关数据库。我们从ImmPort官网 (https://www.immport.org/shared/genelists) 获取免疫基因列表。
1.2. 差异基因筛选和GO、KEGG注释
将TCGA-SKCM转录组数据count值矩阵导入R软件,使用R软件包“DESeq2”对TCGA-SKCM数据集中转移和原发SKCM进行差异表达基因(differentially expressed genes,DEGs)的筛选。首先通过设置参数为75%的样本里count值大于10来过滤低表达基因,然后使用校正P<0.05和Log2差异倍数(fold change,FC)的绝对值>1作为筛选标准获得DEGs。
将分析得到的DEGs导入到R软件包“clusterProfiler”进行基因本体论(Gene Ontology,GO)[13]分析和京都基因与基因组百科全书(Kyoto Encyclopedia of Genes and Genomes,KEGG)[14]通路富集。GO参数设置为 P<0.05,Q<0.25;KEGG参数设置基因数为5~500,其余参数与GO分析一致。最后选择排名前10的GO和KEGG通路进行可视化。
1.3. 预后模型建立
先将分析得到的DEGs与免疫基因列表取交集,进一步将交集中的基因在TCGA-SKCM数据集对总生存期(overall survival,OS)进行单因素Cox回归分析。将单因素Cox回归分析中P<0.05的基因定义为预后相关基因用于后续分析。因LASSO方法可以通过构造一个惩罚函数得到一个较为精炼的模型,故进一步使用LASSO方法对预后相关基因进行变量筛选。最后使用逐步回归Cox方法建立预后模型,通过加权Cox回归系数计算每个患者的风险评分。公式如下:
在训练队列TCGA-SKCM数据集中对使用风险评分预测1、3、5和10年的OS的准确性进行测试,并在验证队列GSE54467和GSE65904数据集中对模型结果进行验证。使用R软件包“survivalROC”计算时间依赖的受试者操作特征(receiver operating characteristic,ROC)曲线下的面积(area under curve,AUC),用以评估预后模型预测能力的准确性。
1.4. 生存分析
从训练队列和验证队列的临床数据中提取OS及状态信息,结合建模基因表达谱数据和所构建的预后模型,使用R软件包“survminer”中的“surv_cutpoint”函数确定最佳的风险评分截断值,将患者划分为高风险评分和低风险评分人群,最后使用Kaplan-Meier方法绘制生存曲线。
1.5. 肿瘤微环境分析
CIBERSORT是一个R/网页版工具,可以通过使用基因表达数据矩阵去卷积方法评估混合细胞群中免疫细胞的浸润丰度。为探讨构建的预后模型与免疫细胞浸润水平的相关性,采用CIBERSORT工具在TCGA-SCKM数据集中评估22种免疫细胞的浸润丰度。基于表达估计恶性肿瘤组织的基质细胞和免疫细胞(Estimation of STromal and Immune cells in MAlignant Tumor tissues using Expression data,ESTIMATE)算法可以根据表达数据估计肿瘤样本的基质分数和免疫分数,用于推断肿瘤微环境中基质和免疫细胞比例[15]。采用追踪肿瘤免疫表型(tracking tumor immunophenotype,TIP)[16]评估肿瘤免疫周期。另外,从以往研究[17]中获得20个具有治疗潜力的抑制性免疫检查点,并从R软件包“IOBR”[18]获取17条已经明确定义的肿瘤微环境相关通路进行更多的肿瘤免疫微环境分析。
1.6. SKCM皮损与正常皮肤组织RNA分离及实时 反转录聚合酶链反应
本研究涉及的人体组织标本均获受试者的知情同意书。SKCM皮损组织取自中南大学湘雅医院皮肤科湘雅医院皮肤科(以下简称“我科”)就诊的SKCM患者,正常对照来自在门诊手术室修剪的正常皮肤组织。组织样本的获取及实验方案均通过中南大学湘雅医院医学伦理委员会的批准(审批号:201905-r19013)。组织用TRIzol试剂(美国Invitrogen公司)分离得到RNA。采用反转录试剂盒(美国Qiagen公司)将RNA反转录合成cDNA,使用Bio-Rad CFX96实时荧光定量PCR系统进行实时反转录聚合酶链反应(real-time reverse transcription PCR,real-time RT-PCR)。靶基因mRNA的表达标化为相对于内参基因甘油醛-3-磷酸脱氢酶(glyceraldehyde-3-phosphate dehydrogenase,GAPDH)的表达,计算公式如下:ΔΔCt=(Ct靶基因-Ct内参基因)-(Ct靶基因-Ct内参基因)。所用引物的序列见表1。
表 1.
实时反转录聚合酶链反应引物序列
Table 1 Sequences of real-time reverse transcription PCR primers
| Genes | Forward primer(5'→3') | Reverse primer(5'→3') |
|---|---|---|
| CRABP2 | TTGAGGAGCAGACTGTGGATGG | GTTCTCTGGTCCACGAGGTCTT |
| LIF | AGATCAGGAGCCAACTGGCACA | GCCACATAGCTTGTCCAGGTTG |
| BMP6 | CCGACAACAGAGTCGTAATCGC | CTGCCATCCCAGGTCTTGGAAA |
| KLRD1 | GAGCCAGCATTTACTCCAGGAC | GCACAGAGATGCCGACTTTCGT |
| GAPDH | ATGGTGAAGGTCGGTGTGA | AATCTCCACTTTGCCACTGC |
CRABP2: Cellular retinoic acid binding protein 2; LIF: Leukemia inhibitory factor; BMP6: Bone morphogenetic protein 6; KLRD1: Killer cell lectin like receptor D1; GAPDH: Glyceraldehyde-3-phosphate dehydrogenase.
1.7. SKCM皮损与正常皮肤组织蛋白质印迹检测
为探究预后模型中关键基因在本研究SKCM与正常对照患者皮肤组织中的蛋白表达差异,使用放射免疫沉淀分析(radio-immunoprecipitation assay,RIPA)裂解缓冲液(上海碧云天生物技术有限公司)进行皮肤组织的裂解。二喹啉甲酸(bicinchoninic acid,BCA)蛋白检测试剂盒(上海碧云天生物技术有限公司)用于测定蛋白质浓度。根据目标蛋白质分子量大小配置8%~12%的十二烷基硫酸钠聚丙烯酰胺凝胶电泳(sodium dodecyl-sulfate polyacrylamide gel electrophoresis,SDS-PAGE)凝胶进行电泳,随后通过聚偏二氟乙烯(polyvinylidene fluoride,PVDF)膜(美国Millipore公司)进行转膜。转膜后使用5%脱脂牛奶在室温下封闭1 h。将膜置于含有一抗人细胞维甲酸结合蛋白2(cellular retinoic acid binding protein 2,CRABP2)[1꞉1 000,武汉爱博泰克(ABclonal)生物科技有限公司]、白血病抑制因子(leukemia inhibitory factor,LIF)[1꞉1 000,武汉爱博泰克(ABclonal)生物科技有限公司]、杀伤细胞凝集素样受体D1(killer cell lectin like receptor D1,KLRD1)[1꞉1 000,武汉爱博泰克(ABclonal)生物科技有限公司]、GAPDH(1꞉3 000,美国Proteintech Group公司)的封闭盒孵育(4 ℃,过夜)。隔日使用抗-HRP-兔IgG(1꞉3 000,美国Proteintech Group公司)进行二抗孵育。最后通过Odyssey Li-Cor系统(美国LI-COR公司)对条带进行统计分析。
1.8. 统计学处理
采用GraphPad Prism 8.0(美国GraphPad公司)生成相关数据的图表并进行分析,计量资料采用均数±标准差( ±s)表示。2个独立样本之间的平均值比较采用t检验,采用Kaplan-Meier法比较2个亚组的总生存率,采用Wilcoxon检验比较两组间差异,Spearman相关分析用于评估风险评分和免疫特征之间的相关性。P<0.05为差异具有统计学意义。
2. 结 果
2.1. DEGs筛选及GO分析和KEGG通路富集结果
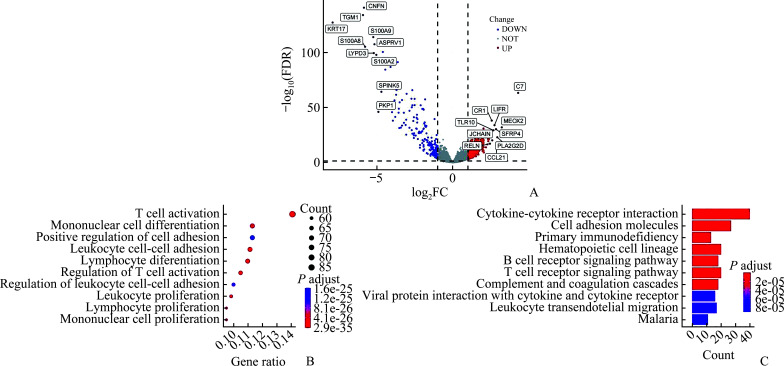
筛选的数据集包括103例原发性SKCM和369例转移性SKCM肿瘤样本(TCGA-SKCM数据集)。根据其转录组测序count值,通过R软件包“DESeq2”进行DEGs,共获得DEGs 710个,其中上调的DEGs 523个,下调的DEGs 187个(校正P<0.05,|Log2FC|>1;图1A)。
图1.
TCGA-SCKM数据集DEGs筛选及GO分析和KEGG通路富集结果
Figure 1 Screening of DEGs in TCGA-SCKM dataset and the results of GO enrichment analysis and KEGG pathway enrichment
A: Comparison of up-regulated and down-regulated genes in the TCGA-SKCM database between primary and metastatic melanoma; B: Analysis of differential genes using GO enrichment analysis; C: KEGG pathway enrichment analysis of DEGs. TCGA-SCKM:The Cancer Genome Atlas-Skin cutaneous melanoma; DEGs: Differentially-expressed genes; GO:Gene Ontology; KEGG: Kyoto Encyclopedia of Genes and Genomes; FC: Fold change; FDR: False discovery rate.
GO分析结果主要包括:T细胞的活化、单核细胞分化、细胞黏附的正向调节、白细胞的细胞间黏附、淋巴细胞分化等(图1B)。KEGG通路富集分析结果主要包括:细胞因子间受体的相互作用、细胞黏附分子、原发性免疫缺陷、造血细胞谱系、T细胞受体通路等(图1C)。
2.2. 免疫相关预后模型的建立
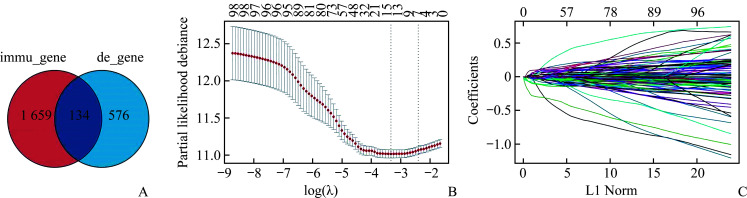
单因素Cox回归分析显示:在训练队列TCGA-SCKM有98个与预后显著相关且P值小于0.05的DEGs。通过LASSO方法和逐步回归进一步过滤变量,最终筛选出CRABP2、LIF、BMP6和KLRD1基因来构建预后模型(图2)。预后风险评分计算公式为:风险评分=0.151 259 3×CRABP2表达量-0.110 981 4×LIF表达量-0.157 720 7×BMP6表达量-0.518 892 8×KLRD1表达量。
图2.
构建免疫相关预后模型
Figure 2 Construction of immune-related prognostic model
A: Genetic interaction between immune-related and differential gene sets; B: Analysis of LASSO-modeled prognostic genes; C: Cross-validation of LASSO-modeled prognostic genes.
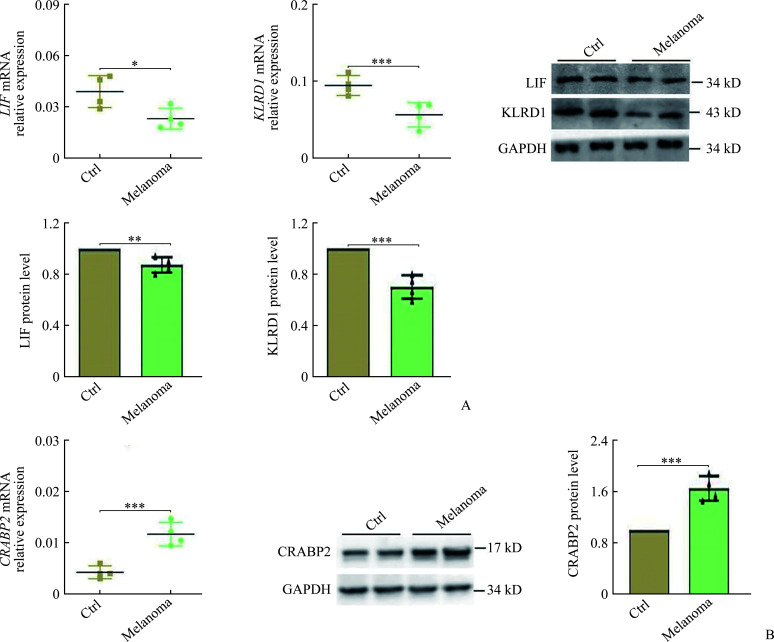
2.3. 在SKCM组织样本中验证预后模型相关基因的mRNA和蛋白表达水平
Real-time RT-PCR和蛋白质印迹法结果显示:与正常皮肤组织相比,LIF和KLRD1的mRNA和蛋白质表达水平在SKCM皮损组织中均下调(均P<0.05,图3A),CRABP2的mRNA和蛋白质表达水平在SKCM皮损组织中均上调(均P<0.001,图3B)。
图3.
在皮肤黑色素瘤组织样本中验证预后模型相关基因的mRNA和蛋白水平表达
Figure 3 mRNA and protein expression of prognostic model-associated genes in skin cutaneous melanoma tissue samples
A: Validation of mRNA and protein levels of LIF and KLRD1 in melanoma lesions and control (Ctrl) skin using real-time reverse transcription PCR and Western blotting (n=5, one sample excluded due to tissue degradation); B: Comparison of CRABP2 mRNA and protein levels for melanoma lesions and control skin (n=5, one sample excluded due to tissue degradation). Data are presented as mean±standard deviation, *P<0.05, **P<0.01, ***P<0.001.
2.4. 验证预后模型
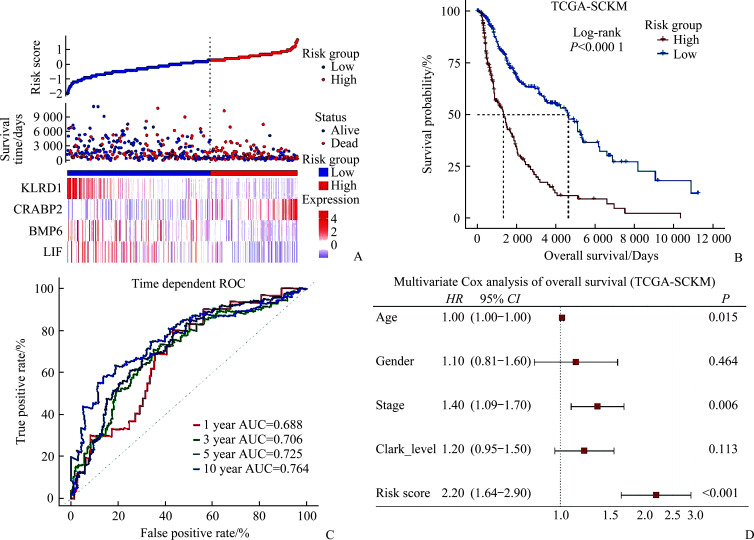
在训练队列中计算每位SKCM患者的风险评分,选择OS大于0的患者(TCGA-SCKM训练队列数据集),共计458例患者被纳入。使用“surv_cutpoint”函数计算得到最佳风险评分截断值,将这458名患者分为高风险评分组210例和低风险评分组248例(图4A)。Kaplan-Meier生存分析显示:高风险评分组的OS率显著低于低风险评分组(图4B)。时间依赖的ROC显示:模型1、3、5及10年的AUC值分别为0.688、0.706、0.725及0.764(图4C)。多因素Cox回归分析显示风险评分是独立的预后危险因素(图4D)。
图4.
训练队列TCGA-SCKM
Figure 4 Training cohort of TCGA-SCKM
A: Classification of high- and low-risk groups according to prognostic risk models for the training cohort; B: Survival analysis using prognostic hazard models for the training cohort of TCGA-SCKM; C: Analysis of TCGA-SCKM training cohort survival using prognostic risk models at 1, 3, 5, 10, and 15 years; D: Multivariate Cox analysis indicating that risk score is an independent predictor of training cohort survival. TCGA-SCKM: The Cancer Genome Atlas-skin cutaneous melanoma; AUC: Area under the curve.
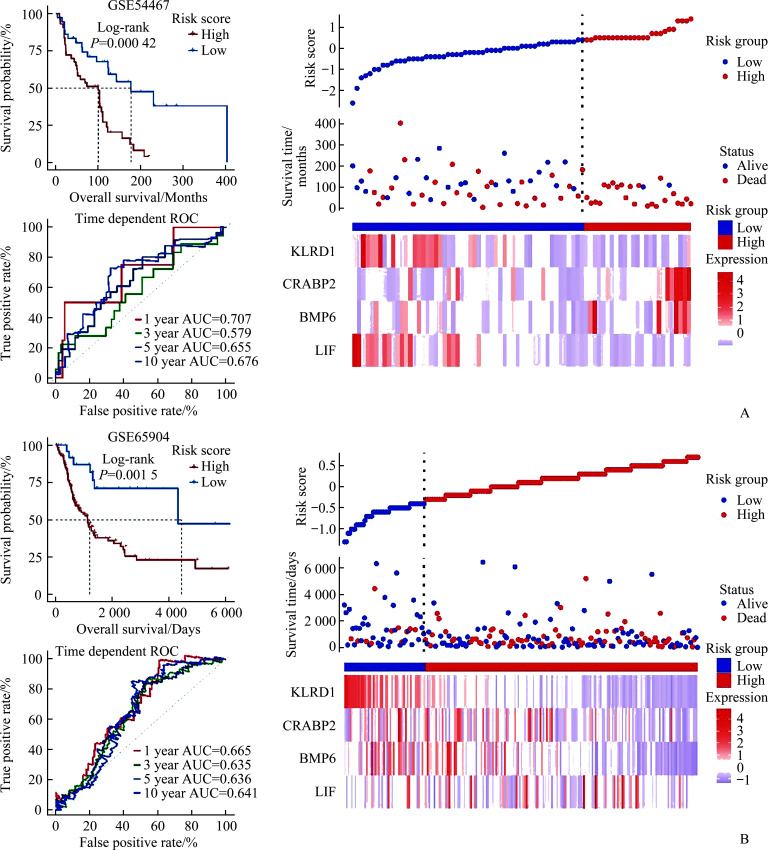
在2个转移性SKCM队列中,GSE54467数据集中,高、低风险评分组OS率差异有统计学意义(P=0.000 42,图5A),1、3、5及10年的AUC值分别为0.707、0.579、0.655及0.676(图5A),GSE54467数据集整体风险模型可视化见图5A。在GSE65904数据集中,高、低风险评分组OS差异有统计学意义(P=0.001 5,图5B),1、3、5及10年的AUC值分别为0.665、0.635、0.636及0.641(图5B),GSE54467数据集整体风险模型可视化见图5B。
图5.
Figure 5 Verification cohort of GSE54467 and GSE65904
A: With the prognostic hazard model, the Kaplan-Meier plot shows the survival probability of the validation cohort GSE544667, ROC curves are shown for survival analyses using 1, 3, 5, and 10-year time-dependent risk models in the validation cohort GSE544667, and prognostic risk models for high- and low-risk groups is validated in GSE544667. B: Kaplan-Meier plot shows the survival probability of the validation cohort GSE65904 using the prognostic risk model. ROC curves are shown for survival analyses using 1, 3, 5, and 10-year time-dependent risk models in the validation cohort GSE65904, and prognostic risk model for high- and low-risk groups is validated in GSE65904.
2.5. 预后模型与肿瘤免疫微环境的相关性
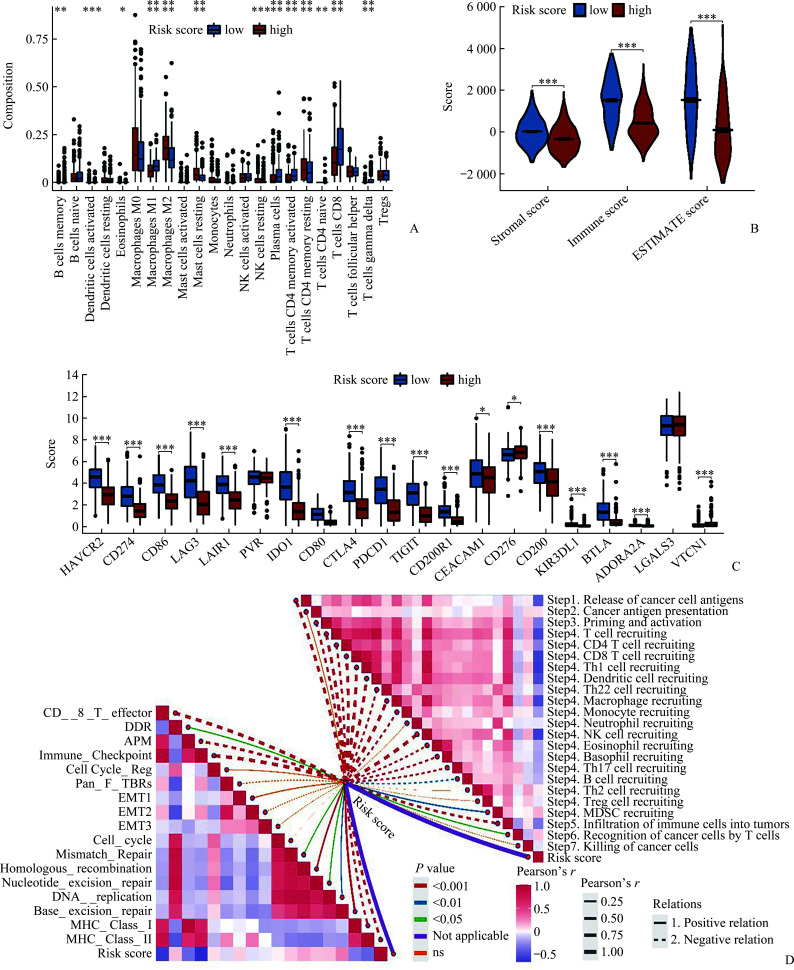
使用CIBERSORT工具计算22种免疫细胞的相对浸润水平,发现在高、低风险评分2组间,大部分的免疫细胞浸润差异有统计学意义(均P<0.01,附图1A,https://doi.org/10.11817/j.issn.1672-7347.2023.230069F1)。其中值得关注的是CD8+ T细胞的浸润水平在高风险组中显著低于低风险组(P<0.001),而高风险评分组中M2型巨噬细胞的浸润水平显著高于低风险组(P<0.001)。高风险评分组表现出更低的免疫评分、基质评分和ESTIMATE评分(附图1B),同时高风险评分组大部分的免疫检查点mRNA表达水平显著低于低风险评分组(附图1C)。从风险评分与肿瘤免疫周期的相关性分析中可以看出,风险评分与大部分的肿瘤免疫周期呈显著的负相关(附图1D)。风险评分与肿瘤微环境通路的相关性分析显示:风险评分与免疫检查点、CD8效应T细胞、MHCⅠ类分子、MHC II类分子、抗原处理呈递机制(antigen processing and presentation mechanism,APC)呈显著负相关(P<0.01,附图1D)。
2.6. 预后模型与肿瘤免疫治疗效果的相关性
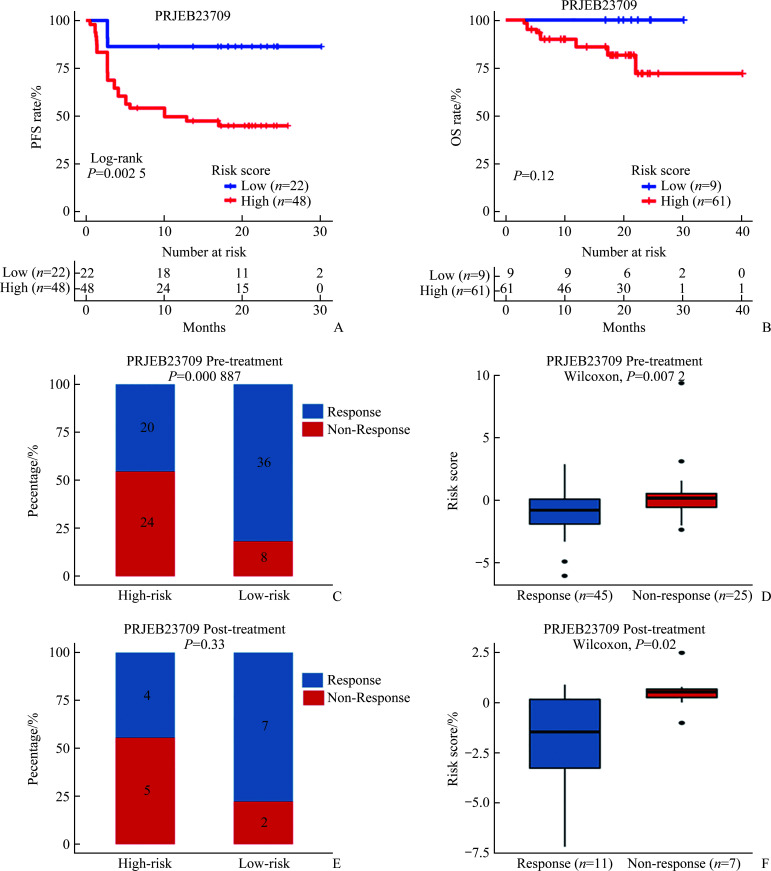
在接受免疫治疗的PRJEB23709数据集中研究风险评分预测患者对ICI治疗反应的能力,发现风险评分较高的患者相较于风险评分较低的患者有更短的无进展生存期(P=0.002 5,Log-rank检验;附图2A,https://doi.org/10.11817/j.issn.1672-7347.2023.230069F2)和OS(P=0.12,Log-rank检验;附图2B);在PRJEB23709数据集88例ICI治疗前取样的患者中,高风险评分的患者对PD-1治疗无反应的占比明显高于低风险评分的患者(24/44 vs 8/44;P=0.000 887,χ2检验;附图2C)。此外,在ICI治疗后取样的患者中也观察到一致的现象(5/9 vs 2/9;Fisher精确概率法:P=0.33;附图2E)。与此同时,无论在治疗前取样的患者中还是在治疗后取样的患者中,ICI治疗抵抗的患者相较于敏感的患者有更高的风险评分(附图2D~2F)。
3. 讨 论
本研究首先通过分析TCGA数据库中原发性与转移性SKCM的DEGs及一系列富集分析发现DEGs主要与肿瘤免疫微环境相关。然后与免疫基因取交集,并使用LASSO及逐步回归分析法,用CRABP2、LIF、BMP6和KLRD1基因构建了一个SKCM的预后模型;在训练队列与多个验证队列中取得一致的结果,并通过模型计算的风险评分评估了SKCM患者的肿瘤免疫微环境状态,揭示了风险评分与免疫细胞浸润、ESTIMATE评分、免疫检查点mRNA表达水平、肿瘤免疫周期以及肿瘤微环境通路之间的联系。这些结果为指导SKCM的临床治疗及预后预测提供了新线索。结合目前的文献报道及本研究多数据集和临床样本的初步验证结果表明:在预后模型中关键预后标志物有一定的稳定性及可靠性,预后模型在SKCM免疫治疗的效果预测方面具有重要的潜在应用价值。
本研究结果显示:风险评分模型中高风险评分的SKCM患者免疫治疗预后较差,并且大部分免疫检查点的表达水平以及CD8+效应T细胞浸润水平显著低于低风险评分的患者。免疫检查点的表达水平和CD8+效应T细胞浸润水平通常被认为是预测免疫治疗效果的生物标志物[19-20]。M1型巨噬细胞主要塑造抗肿瘤的免疫微环境,而M2型巨噬细胞主要塑造抑制性的肿瘤免疫微环境[21]。在预后模型与肿瘤免疫微环境的相关性分析中,高风险评分人群的M1巨噬细胞表现出低浸润水平,而M2型巨噬细胞表现出高浸润水平,提示高风险评分可能与抑制性的肿瘤免疫微环境有关。同时,高风险评分与多条免疫相关的通路呈负相关,进一步表明高风险评分与抑制性肿瘤免疫微环境存在联系。根据报道[22-25],CRABP2、LIF、BMP6和KLRD1基因也可通过影响细胞周期或免疫细胞募集等影响肿瘤免疫微环境。另外,本研究发现高风险评分的患者免疫治疗效果更差,无进展生存期或OS率均显著短于或低于低风险评分的患者。进一步表明所建立的风险评分模型可以被视为免疫治疗反应的预测标志物。
笔者建立预后模型的4个免疫相关基因已被证实与SKCM相关[26-29]。其中,CRABP2可以独立作为SKCM的危险预后因素,高表达CRABP2的SKCM OS率更低[26]。LIF与p21信号通路级联,作用于转化生长因子(transforming growth factor,TGF)-β 的下游以调节SKCM肿瘤细胞周期停滞和细胞死亡[27]。在SKCM小鼠模型中BMP6缺乏导致肿瘤发生显著延迟和肿瘤进展减慢[28]。KLRD1,也被称为CD94,是自然杀伤(natural killer,NK)细胞上表达的抗原。CD94+细胞中超过95%共表达CD8,CD8+细胞群中CD94表达的百分比在5%至20%之间,在转移性SKCM中表达较高[29]。临床样本验证结果表明CRABP2、LIF及KLRD1基因在SKCM组织与正常组织中存在显著差异表达,进一步提示模型中关键基因与SKCM密切相关。
目前肿瘤治疗的研究取得了很大的进展,免疫治疗为患者提供了新的治疗选择。但对于肿瘤患者预后的预测以及免疫治疗潜在获益人群的筛选,单一的基因表达或临床指标往往不够。基于高通量测序大数据的生物信息学分析,能够更好地寻找可靠的生物标志物。本研究首次提出基于CRABP2、LIF、BMP6和KLRD1的预测模型,为SKCM治疗的预后生物标志物的探索提供了新的思路,通过进一步分析及验证,证实预后模型具有较强预测能力。总的来说,本研究为改善SKCM的免疫治疗结果提供了新的可能性。然而,本研究也有一些局限性:为生物信息学分析,需要更多的前瞻性研究来验证结论;没有进行深入的体内外验证,需通过体内外细胞生物学实验,在分子机制层面更深入地探索并证实此预后模型。今后,除预后模型中的关键分子外,还应关注外周血和皮肤组织中其他的生物标志,以实现对SKCM免疫治疗的系统评估。
附录.
附图1. 风险评分与肿瘤免疫微环境的相关性Supplementary Figure 1 Correlation of risk score and tumor immune microenvironment .
A: Comparison of 22 types of immune cells infiltration between the high- and low-risk groups of the TCGA-SCKM cohort; B: ESTIMATE algorithm comparison of immune infiltration scores in high- and low-risk groups; C: Relative expression levels of 20 immune checkpoint mRNAs in the high- and low-risk groups; D: Tumor microenvironment pathway scores and tumor immune cycle scores in the high- and low-risk groups.
附图2. 风险评分预测免疫治疗效果Supplementary Figure 2 Risk score in efficacy prediction of immunotherapy .
A and B: Kaplan-Meier curves show progression-free survival (PFS) rate (A) and overall survival (OS) rate (B) in the high-risk (red) and low-risk (blue) groups before ICI treatment of melanoma ICI cohorts (PRJEB23709). P<0.05 in the two-sided log-rank test was considered statistically significant. C and E: Proportion of patients with different responses to immunotherapy of pre-treatment (C, the Chi-squared test) or post-treatment (E, Fisher test) samples in melanoma ICI cohorts (PRJEB23709). Response, including complete response (CR) and partial response (PR); Non-response, including stable disease (SD) and progressive disease (PD). D and F: Difference in the risk score between R (responders) and NR (non-responders) patients of pre-treatment (D) or post-treatment (F) samples in melanoma ICI cohorts (PRJEB23709). P value was determined by Wilcoxon rank-sum test. ICI: Immune-checkpoint inhibitor.
基金资助
国家自然科学基金(82073018);湖南省自然科学基金(2022JJ30189);深圳市科技创新委员会项目(JCYJ20210324113001005);湖南省高校教学改革研究项目(HNJG-2021-1120)。
This work was supported by the National Natural Science Foundation (82073018), the Natural Science Foundation of Hunan Province (2022JJ30189), Project of Shenzhen Science, the Technology and Innovation Commission (JCYJ20210324113001005), and the Teaching Reform Research Project of Universities in Hunan Province (HNJG-2021-1120), China.
利益冲突声明
作者声称无任何利益冲突。
作者贡献
吴猛 实验操作,数据采集及统计分析,论文撰写与修改;汪政、张江林 实验设计和指导,论文修改。所有作者阅读并同意最终的文本。
原文网址
http://xbyxb.csu.edu.cn/xbwk/fileup/PDF/202305671.pdf
参考文献
- 1. Bray F, Ferlay J, Soerjomataram I, et al. Global cancer statistics 2018: GLOBOCAN estimates of incidence and mortality worldwide for 36 cancers in 185 countries[J]. CA Cancer J Clin, 2018, 68(6): 394-424. 10.3322/caac.21492. [DOI] [PubMed] [Google Scholar]
- 2. Spagnolo F, Boutros A, Tanda E, et al. The adjuvant treatment revolution for high-risk melanoma patients[J]. Semin Cancer Biol, 2019, 59: 283-289. 10.1016/j.semcancer.2019.08.024. [DOI] [PubMed] [Google Scholar]
- 3. Binnewies M, Roberts EW, Kersten K, et al. Understanding the tumor immune microenvironment (TIME) for effective therapy[J]. Nat Med, 2018, 24(5): 541-550. 10.1038/s41591-018-0014-x. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 4. De Jaeghere EA, Denys HG, De Wever O. Fibroblasts fuel immune escape in the tumor microenvironment[J]. Trends Cancer, 2019, 5(11): 704-723. 10.1016/j.trecan.2019.09.009. [DOI] [PubMed] [Google Scholar]
- 5. Fortes C, Mastroeni S, Mannooranparampil TJ, et al. Tumor-infiltrating lymphocytes predict cutaneous melanoma survival[J]. Melanoma Res, 2015, 25(4): 306-311. 10.1097/CMR.0000000000000164. [DOI] [PubMed] [Google Scholar]
- 6. Maibach F, Sadozai H, Seyed Jafari SM, et al. Tumor-infiltrating lymphocytes and their prognostic value in cutaneous melanoma[J]. Front Immunol, 2020, 11: 2105. 10.3389/fimmu.2020.02105. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 7. Mao XQ, Xu J, Wang W, et al. Crosstalk between cancer-associated fibroblasts and immune cells in the tumor microenvironment: new findings and future perspectives[J]. Mol Cancer, 2021, 20(1): 131. 10.1186/s12943-021-01428-1. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 8. Gide TN, Wilmott JS, Scolyer RA, et al. Primary and acquired resistance to immune checkpoint inhibitors in metastatic melanoma[J]. Clin Cancer Res, 2018, 24(6): 1260-1270. 10.1158/1078-0432.CCR-17-2267. [DOI] [PubMed] [Google Scholar]
- 9. Suresh K, Naidoo J, Lin CT, et al. Immune checkpoint immunotherapy for non-small cell lung cancer: benefits and pulmonary toxicities[J]. Chest, 2018, 154(6): 1416-1423. 10.1016/j.chest.2018.08.1048. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 10. Krasniqi E, Barchiesi G, Pizzuti L, et al. Immunotherapy in HER2-positive breast cancer: state of the art and future perspectives[J]. J Hematol Oncol, 2019, 12(1): 111. 10.1186/s13045-019-0798-2. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 11. 王丽惠, 黄程辉. 免疫检查点抑制剂在结直肠癌中的应用[J]. 中南大学学报(医学版), 2021, 46(8): 894-899. 10.11817/j.issn.1672-7347.2021.200872 [DOI] [PMC free article] [PubMed] [Google Scholar]; WANG Lihui, HUANG Chenghui. Application of immune checkpoint inhibitors in colorectal cancer[J]. Journal of Central South University. Medical Science, 2021, 46(8): 894-899. 10.11817/j.issn.1672-7347.2021.200872 [DOI] [PMC free article] [PubMed] [Google Scholar]
- 12. O’Donnell JS, Teng MWL, Smyth MJ. Cancer immunoediting and resistance to T cell-based immunotherapy[J]. Nat Rev Clin Oncol, 2019, 16(3): 151-167. 10.1038/s41571-018-0142-8. [DOI] [PubMed] [Google Scholar]
- 13. Ashburner M, Ball CA, Blake JA, et al. Gene ontology: tool for the unification of biology. The Gene Ontology Consortium[J]. Nat Genet, 2000, 25(1): 25-29. 10.1038/75556. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 14. Kanehisa M, Goto S. KEGG: Kyoto encyclopedia of genes and genomes[J]. Nucleic Acids Res, 2000, 28(1): 27-30. 10.1093/nar/28.1.27. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 15. Yoshihara K, Shahmoradgoli M, Martínez E, et al. Inferring tumour purity and stromal and immune cell admixture from expression data[J]. Nat Commun, 2013, 4: 2612. 10.1038/ncomms3612. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 16. Xu LW, Deng CY, Pang B, et al. TIP: a web server for resolving tumor immunophenotype profiling[J]. Cancer Res, 2018, 78(23): 6575-6580. 10.1158/0008-5472.CAN-18-0689. [DOI] [PubMed] [Google Scholar]
- 17. Auslander N, Zhang G, Lee JS, et al. Robust prediction of response to immune checkpoint blockade therapy in metastatic melanoma[J]. Nat Med, 2018, 24(10): 1545-1549. 10.1038/s41591-018-0157-9. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 18. Zeng DQ, Ye ZL, Shen RF, et al. IOBR: multi-omics immuno-oncology biological research to decode tumor microenvironment and signatures[J]. Front Immunol, 2021, 12: 687975. 10.3389/fimmu.2021.687975. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 19. Raskov H, Orhan A, Christensen JP, et al. Cytotoxic CD8+ T cells in cancer and cancer immunotherapy[J]. Br J Cancer, 2021, 124(2): 359-367. 10.1038/s41416-020-01048-4. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 20. Gubin MM, Zhang XL, Schuster H, et al. Checkpoint blockade cancer immunotherapy targets tumour-specific mutant antigens[J]. Nature, 2014, 515(7528): 577-581. 10.1038/nature13988. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 21. Boutilier AJ, Elsawa SF. Macrophage polarization states in the tumor microenvironment[J]. Int J Mol Sci, 2021, 22(13): 6995. 10.3390/ijms22136995. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 22. Jauch AS, Wohlfeil SA, Weller C, et al. Lyve-1 deficiency enhances the hepatic immune microenvironment entailing altered susceptibility to melanoma liver metastasis[J]. Cancer Cell Int, 2022, 22(1): 398. 10.1186/s12935-022-02800-x. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 23. Huber M, Brehm CU, Gress TM, et al. The immune microenvironment in pancreatic cancer[J]. Int J Mol Sci, 2020, 21(19): 7307. 10.3390/ijms21197307. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 24. Wang Q, Vattai A, Vilsmaier T, et al. Immunogenomic identification for predicting the prognosis of cervical cancer patients[J]. Int J Mol Sci, 2021, 22(5): 2442. 10.3390/ijms22052442. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 25. Datta RR, Schran S, Persa OD, et al. Post-transplant malignancies show reduced T-cell abundance and tertiary lymphoid structures as correlates of impaired cancer immunosurveillance[J]. Clin Cancer Res, 2022, 28(8): 1712-1723. 10.1158/1078-0432.CCR-21-3746. [DOI] [PubMed] [Google Scholar]
- 26. Yan JY, Wu XW, Yu JY, et al. Prognostic role of tumor mutation burden combined with immune infiltrates in skin cutaneous melanoma based on multi-omics analysis[J]. Front Oncol, 2020, 10: 570654. 10.3389/fonc.2020.570654. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 27. Humbert L, Ghozlan M, Canaff L, et al. The leukemia inhibitory factor (LIF) and p21 mediate the TGFβ tumor suppressive effects in human cutaneous melanoma[J]. BMC Cancer, 2015, 15: 200. 10.1186/s12885-015-1177-1. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 28. Stieglitz D, Lamm S, Braig S, et al. BMP6-induced modulation of the tumor micro-milieu[J]. Oncogene, 2019, 38(5): 609-621. 10.1038/s41388-018-0475-x. [DOI] [PubMed] [Google Scholar]
- 29. Vetter CS, Straten PT, Terheyden P, et al. Expression of CD94/NKG2 subtypes on tumor-infiltrating lymphocytes in primary and metastatic melanoma[J]. J Invest Dermatol, 2000, 114(5): 941-947. 10.1046/j.1523-1747.2000.00958.x. [DOI] [PubMed] [Google Scholar]